Algoritmos contra doenças
Ferramenta de bioinformática patenteada pela UFMG pode ajudar no desenvolvimento de medicamentos
Por Luana Macieira
04/02/26, às 08h00
A tecnologia sempre foi uma aliada nos tratamentos de saúde, com presença cada vez mais constante nas pesquisas e estudos do campo das ciências biológicas. Com o objetivo de simular a ligação de moléculas com potencial farmacológico, um grupo de pesquisadores da UFMG desenvolveu o POTTER (peptide optmization tool for enhanced receptor-binding). A inovação é uma ferramenta computacional baseada em algoritmos genéticos para otimização de peptídeos com foco na inibição de proteínas-alvo. O projeto parte do trabalho da pesquisadora Ana Paula Abreu, que cursou o doutorado em Bioinformática na UFMG, sob orientação do professor Adriano de Paula Sabino, do Departamento de Análises Clínicas e Toxicológicas da Faculdade de Farmácia.
Durante a pandemia da covid-19, liderado pelo professor Adriano de Paula Sabino e pela professora Raquel Minardi, do Departamento de Ciência da Computação (DCC), o grupo de pesquisadores desenvolveu um algoritmo que possibilita o planejamento e a simulação de interações de moléculas com potencial para inibir a interação do vírus Sars-CoV-2, causador da covid-19, com as células do hospedeiro.
Essas moléculas são os peptídeos. Eles são compostos de resíduos de aminoácidos de duas a 50 sequências, formados por ligações que desempenham papéis cruciais nos seres vivos, como a sinalização celular e a modulação imunológica. Na terapêutica, os peptídeos apresentam diversas características promissoras se comparados a pequenas moléculas e a proteínas terapêuticas, incluindo a alta compatibilidade estrutural com proteínas-alvo e a capacidade de romper interfaces proteína-proteína. Além disso, são altamente seletivos e eficazes, ao mesmo tempo que seguros e bem tolerados.
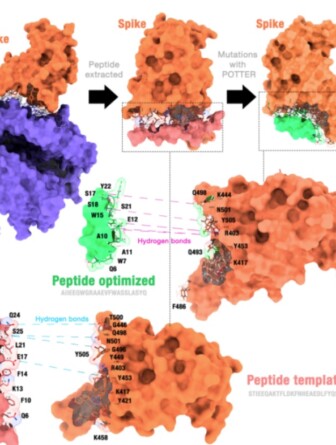
O algoritmo criado pelos pesquisadores simula o desenho dessas moléculas, de forma a impedir a interação da proteína spike do vírus da covid-19 com o alvo na célula hospedeira – a enzima conversora de angiotensina II (ACE2).
Sem ligação, sem infecção
“A técnica é a seguinte: à época do boom da pandemia, descobriu-se que o vírus causador da covid-19 se ligava a uma proteína específica do corpo humano chamada enzima conversora de angiotensina II (ACE2). Por meio do algoritmo criado por nós, podemos desenvolver peptídeos com potencial de impedir a ligação do Sars-CoV-2 (spike) ao alvo (ACE2), que é a porta de entrada no hospedeiro. Sem essa ligação, não há infecção viral”, explica o professor Sabino.
O POTTER avalia e aprimora os peptídeos com base em critérios como o perfil de interação das moléculas, a porcentagem de resíduos em contato no local de interação e a pontuação de ancoragem. Após a análise da melhor sequência inicial com potencial inibidor, o grupo realizou uma série de simulações, gerando uma infinidade de sequências possíveis. Nesse processo, foram selecionadas aquelas que apresentaram as interações mais favoráveis.
“As 10 melhores sequências de peptídeos foram então sintetizadas para validação experimental. Com elas em mãos, pretendemos realizar os testes inibitórios. A intenção é observar se algum desses 10 peptídeos terá a ação esperada in vitro, conforme observado na modelagem in silico – quando os testes são feitos computacionalmente antes de irem para a bancada do laboratório”, diz o professor.
Base replicável
Adriano Sabino acrescenta que a ideia inicial do grupo era empregar o POTTER no estudo de peptídeos que poderiam ser utilizados na fabricação de medicamentos para tratamentos oncológicos. Porém, com o início da pandemia, o estudo acabou direcionado para o Sars-CoV-2.
Apesar de a pesquisa ter sido realizada com foco no vírus da covid-19, o professor destaca que a tecnologia também pode ser aplicada ao desenvolvimento de peptídeos capazes de se ligar a moléculas ou alvos associados a outras doenças. “Todos os passos que realizamos com o vírus da covid-19 podem ser feitos com alvos de outras doenças. A base do nosso estudo é facilmente replicável em outros trabalhos”, diz.
Em relação ao estudo que originou a patente, o professor explica que o próximo passo é desenvolver os testes pré-clínicos in vitro, seguidos dos testes pré-clínicos in vivo. A última fase do estudo, caso confirmada a efetividade nos testes anteriores, é a busca por parceiros da indústria farmacêutica.

“O desenvolvimento do algoritmo se baseia em bioinformática, modelagem computacional e estudo in silico. Isso tudo tem um custo computacional alto, pois precisamos de servidores robustos, de programadores e de engenheiros de software. No futuro, queremos avançar o projeto com o uso de inteligência artificial. É uma maneira de usar a tecnologia como uma aliada nos tratamentos de doenças”, conclui Sabino.
Além do professor Adriano de Paula Sabino, a equipe de pesquisadores que patenteou a tecnologia POTTER (Peptide optimization tool for enhanced receptor-binding) inclui a professora Raquel Cardoso de Melo Minardi, do Departamento de Ciência da Computação (DCC) da UFMG, e os pesquisadores Diego César Batista Mariano, Ana Paula de Abreu e Frederico Chaves Carvalho.
Matéria replicada do site da UFMG: https://www.ufmg.br/comunicacao/noticias/pesquisa-e-inovacao/algoritmos-contra-doencas/









